Обзор базовых возможностей pandas
Contents
Обзор базовых возможностей pandas¶
Продемонстрируем возможности pandas на реальных данных. Возьмём для этих целей данные о коронавирусе от университета Джонса Хопкинса и от Яндекса.
Университет Джонса Хопкинса собирает данные по коронавирусу и хранит их в открытом доступе в репозитории. Среди прочего, в них содержится информация о количестве заболевших, погибших и выздоровевших. Данные хранятся не в типичном для
pandasформате: информация за одни день хранится в столбце, а не в строках, т.е. не в соответствии с форматом панельных данных. Чтобы не усложнять изложение, я составил скрипт, который скачивает таблицы из репозитория, извлекает из них информацию только по России и сохраняет их в панельном формате. Результат работы этого скрипта от 25.11.2021 хранится по ссылке.Яндекс агрегирует информацию о коронавирусе по всей россии и предоставляет возможность скачать их данные. К сожалению, чтобы получить эти данные, нужно проделать куда больше шагов, чем в примере выше (с инструкцией можно ознакомится здесь). Загруженные от 25.11.2021 данные хранятся здесь.
Чтобы автоматизировать сборку этих материалов и сделать её по возможности устойчивой к отсутствию интернета, в скрытой ячейке ниже находится код, который скачивает упомянутые таблицы с GoogleDrive, если они отсутствуют. Хотя стоит упомянуть, что pandas может считывать данные по url.
from itertools import count
import os
import wget
folder = "data"
os.makedirs(folder, exist_ok=True)
base_url = "https://docs.google.com/uc?export=download&id={}"
to_download = [
{
"file_id": "1ds0Qw-5dTxYvNEWxt1neN4Y9fut8Pnx3",
"filename": "CSSE_Russia.csv",
},
{
"file_id": "1Xw-jyEh5TOAvy5L1iT0Cs68ybP8BaEJb",
"filename": "yandex_data.csv",
}
]
for file in to_download:
path = os.path.join(folder, file["filename"])
if not os.path.isfile(path):
download_url = base_url.format(file["file_id"])
wget.download(download_url, out=path)
plotly_save_to = os.path.join("..", "_static", "plotly")
os.makedirs(plotly_save_to, exist_ok=True)
plotly_tmp = os.path.join(plotly_save_to, "tmp.html")
Данные о коронавирусе от университета Джонса Хопкинса¶
Первые 5 строк файла выглядят так.
Date,recovered,deaths,confirmed
2020-01-22,0,0,0
2020-01-23,0,0,0
2020-01-24,0,0,0
2020-01-25,0,0,0
2020-01-26,0,0,0
Видим, что столбец Date хранит даты в стандартном для pandas формате YYYY-MM-DD. Считываем таблицу, указав в качестве индекса этот столбец, не забывая указать pandas, что в нем хранятся даты. С помощью метода info печатаем информацию о таблице.
import pandas as pd
import numpy as np
import os
folder = "covid19"
filename = "CSSE_Russia.csv"
path = os.path.join(folder, filename)
df = pd.read_csv(path, index_col="Date", parse_dates=["Date"])
df.info()
<class 'pandas.core.frame.DataFrame'>
DatetimeIndex: 672 entries, 2020-01-22 to 2021-11-23
Data columns (total 3 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 recovered 672 non-null int64
1 deaths 672 non-null int64
2 confirmed 672 non-null int64
dtypes: int64(3)
memory usage: 21.0 KB
Расшифруем полученный вывод.
Первая строка сообщает, что индексом таблицы является DatetimeIndex в диапазоне от 22 января 2020 гола до 23 ноября 2021 года (формат (YYYY-MM-DD)). Из первых двух строчек можно понять, что в таблице 3 столбца (+ как бы столбец индекса) и 672 строки.
Далее идет информация о каждом столбце таблицы.
recovered— суммарное количество зарегистрированных выздоровевших отcovid19пациентов;deaths— суммарное количество зарегистрированных смертей отcovid19;confirmed— суммарное количество зарегистрированных заболевшихcovid19. Под суммарным количество здесь имеется ввиду то, что на каждую дату в таблице приводятся данные о, например, количестве зарегистрированных больных не только за текущий день, но и за все предшествующие.
Кроме того, из вывода команды info можно понять, что во всех столбцах отсутствуют пропущенные значения NA (672 non-null в 672 строках). Тем не менее это ещё не значит, что в таблице присутствуют данные за все дни в указанном диапазоне. Проверим это пользуясь тем, что можно высчитывать временной интервал между двумя объектами datetime оператором -.
date_range = df.index.max() - df.index.min()
print(date_range.days)
671
Видим, что между 22.01.2020 и 23.11.2021 прошел 671 день, т.е. данные представлены за все дни из этого диапазона, если предположить, что нет двух строк с одной и той же датой. Проявим максимальную осторожность и проверим, что в таблице присутствуют строки для каждого дня в указанном диапазоне. Для этого создадим даты в указанном диапазоне с частотой в день функцией pd.date_range и посмотрим разницу с индексом таблицы.
date_range = pd.date_range(start="22.01.2020", end="23.11.2021", freq="D")
print(f"Созданный диапазон: {date_range}")
difference = date_range.difference(df.index)
print(f"Разница с индексом таблицы: {difference}")
Созданный диапазон: DatetimeIndex(['2020-01-22', '2020-01-23', '2020-01-24', '2020-01-25',
'2020-01-26', '2020-01-27', '2020-01-28', '2020-01-29',
'2020-01-30', '2020-01-31',
...
'2021-11-14', '2021-11-15', '2021-11-16', '2021-11-17',
'2021-11-18', '2021-11-19', '2021-11-20', '2021-11-21',
'2021-11-22', '2021-11-23'],
dtype='datetime64[ns]', length=672, freq='D')
Разница с индексом таблицы: DatetimeIndex([], dtype='datetime64[ns]', freq='D')
C:\Users\qujim\Desktop\python_lectures\venv\lib\site-packages\IPython\core\interactiveshell.py:3369: UserWarning: Parsing '22.01.2020' in DD/MM/YYYY format. Provide format or specify infer_datetime_format=True for consistent parsing.
exec(code_obj, self.user_global_ns, self.user_ns)
C:\Users\qujim\Desktop\python_lectures\venv\lib\site-packages\IPython\core\interactiveshell.py:3369: UserWarning: Parsing '23.11.2021' in DD/MM/YYYY format. Provide format or specify infer_datetime_format=True for consistent parsing.
exec(code_obj, self.user_global_ns, self.user_ns)
Из последней строки вывода видно, что есть данные за каждый день.
Распечатаем таблицу.
df
| recovered | deaths | confirmed | |
|---|---|---|---|
| Date | |||
| 2020-01-22 | 0 | 0 | 0 |
| 2020-01-23 | 0 | 0 | 0 |
| 2020-01-24 | 0 | 0 | 0 |
| 2020-01-25 | 0 | 0 | 0 |
| 2020-01-26 | 0 | 0 | 0 |
| ... | ... | ... | ... |
| 2021-11-19 | 0 | 256669 | 9099253 |
| 2021-11-20 | 0 | 257891 | 9135149 |
| 2021-11-21 | 0 | 259107 | 9170898 |
| 2021-11-22 | 0 | 260319 | 9205431 |
| 2021-11-23 | 0 | 261526 | 9238330 |
672 rows × 3 columns
Вначале идут нули по всем строкам, что объясняется видимо тем, что
Первый официально подтверждённый случай заболевания коронавирусной инфекцией COVID-19 в стационарных учреждениях социальной защиты был зарегистрирован 11 апреля 2020 года. wikipedia.
Так же в столбце по выздоровевшим наблюдаются только нули, что не соответствует действительности, т.е. судя по всему в таблице все же есть отсутствующие значения, которые заполнены нулями, а не np.nan. Визуализируем данные, чтобы а) легче их воспринимать; б) посмотреть на них целиком. Воспользуемся для этого библиотекой plotly, т.к. мы ещё не знаем, что мы ищем и интерактивность может пригодиться.
import plotly.express as px
from IPython.display import display, HTML
fig = px.line(df)
fig.write_html(plotly_tmp) # fig.show()
display(HTML(plotly_tmp))
Из построенного графика можно сделать вывод о том, что данные начали поступать с 12 февраля (несмотря на утверждении в википедии), а начиная с 5 августа 2021 года данные о выздоровевших перестали обновляться.
В данном случае разумно отбросить нулевые строки и заменить пропущенные данные на np.nan.
df = df.loc[np.any(df != 0, axis=1)].copy()
df.loc["2021-08-05":, "recovered"] = np.nan
fig = px.line(df)
fig.write_html(plotly_tmp) # fig.show()
display(HTML(plotly_tmp))
Более или менее понятно, что приблизительно должно выполнятся равенство \(\text{confirmed} = \text{recovered} + \text{deaths}\), т.к. большинство заболевших в итоге выздоравливают или нет. Посмотрим растёт ли невязка этого равенства со временем.
residual = df.confirmed - df.deaths - df.recovered
residual.name = "confirmed - deaths - recovered"
fig = px.line(residual)
fig.write_html(plotly_tmp) # fig.show()
display(HTML(plotly_tmp))
У этого графика наблюдается тенденция роста со временем, что наталкивает на мысль, что
формула не верна;
данные собираются не самым точном образом;
растет средняя продолжительность болезни.
Чтобы оценить динамику, удобно также посмотреть на суточные показатели. Суточный прирост — разница между показателем за выбранный день и за предыдущий день. Чтобы его вычислить, надо посчитать нечто похожее на x[:-1] - x[1:]. Для таких вычислений есть встроенный метод DataFrame.diff (аналогичный метод есть и у pd.Series).
daily_df = df.diff()
fig = px.line(daily_df)
fig.write_html(plotly_tmp) # fig.show()
display(HTML(plotly_tmp))
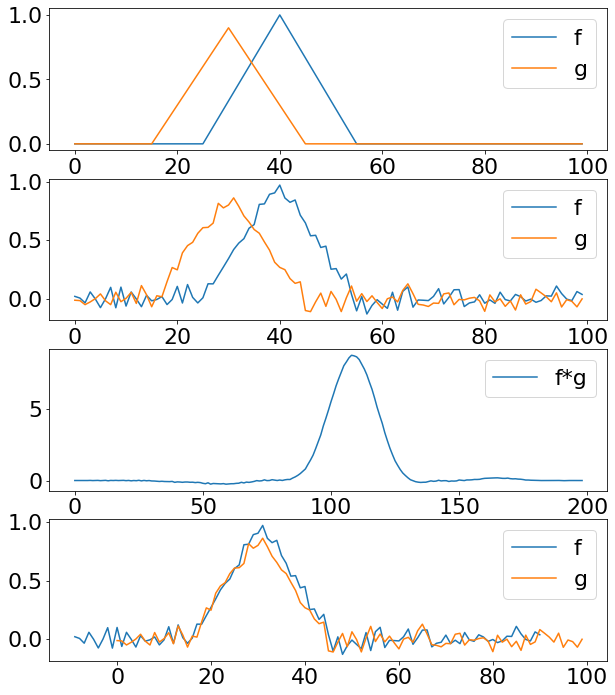
Из этих данных можно попробовать оценить среднюю продолжительность болезни с помощью взаимнокорреляционной функции: за среднюю продолжительность болезни возьмём такое смещение графика confirmed вправо, что он максимально коррелирует с сумой остальных двух. Для этого посчитаем взаимокорреляционную функцию \(f \star g\) величин \(f = \text{confirmed}\) и \(g = \text{recovered} + \text{deaths}\) и возьмём в качестве смещения
Взаимокореляционную функцию (cross corelation) можно вычислить с помощью метода signal.correlate из библиотеки SciPy.
Разница между режимами работы signal.correlate
Если mode='valid', то функция считает корреляцию для всех таких положений g, при которых f не надо дополнять нулями по краям.
Если mode='valid', то функция считает корреляцию для всех положений g, при которых f хотя бы одним элементом накладывается на g.
Если длины f и g равны 5 и 3 соответственно, то результирующий массив в режиме valid выходит размера 3, а в режиме full — 5.
import numpy as np
from scipy import signal
import matplotlib.pyplot as plt
plt.rcParams.update({'font.size': 22})
n = 100
sigma = 0.05
def triangle(center, width, height):
half_width = width / 2
k = height / half_width
def left_side(x):
root = center - half_width
return k * (x - root)
def right_side(x):
root = center + half_width
return -k * (x - root)
x = np.arange(n)
y = left_side(x) * (x < center) + right_side(x) * (x >= center)
return y * (y >= 0)
x = np.arange(n)
f = triangle(40, 30, 1)
g = triangle(30, 30, 0.9)
noisy_f = f + np.random.normal(0, sigma, size=(n, ))
noisy_g = g + np.random.normal(0, sigma, size=(n, ))
corr = signal.correlate(noisy_f, noisy_g, mode="full")
shift = n - np.argmax(corr) - 1
fig, axs = plt.subplots(nrows=4, ncols=1, figsize=(10, 12))
axs[0].plot(x, f, label="f")
axs[0].plot(x, g, label="g")
axs[0].legend()
axs[1].plot(x, noisy_f, label="f")
axs[1].plot(x, noisy_g, label="g")
axs[1].legend()
axs[2].plot(corr, label="f*g")
axs[2].legend()
axs[3].plot(x + shift, noisy_f, label="f")
axs[3].plot(x, noisy_g, label="g")
axs[3].legend()
plt.show(fig)
from scipy import signal
from plotly.subplots import make_subplots
import plotly.graph_objects as go
tmp = daily_df.dropna().copy()
f = tmp.confirmed
g = tmp.recovered + tmp.deaths
f = (f - f.mean()) / f.std()
g = (g - g.mean()) / g.std()
n = len(tmp)
cross_correlation = signal.correlate(f, g, mode="full")
shift = n - np.argmax(cross_correlation) - 1
print(f"Найденный сдвиг: {shift}")
tmp["confirmed shifted on 5"] = tmp.confirmed.shift(shift)
tmp["confirmed shifted on 12"] = tmp.confirmed.shift(12)
tmp["recovered + deaths"] = tmp.recovered + tmp.deaths
target_go = go.Scatter(x=tmp.index, y=tmp["recovered + deaths"], name="recovered + deaths")
correlation_go = go.Scatter(
x=n - np.arange(2 * n - 1) - 1,
y=cross_correlation,
name="cross correlation"
)
fig = make_subplots(rows=2)
# top
fig.append_trace(correlation_go, row=1, col=1)
# bottom
fig.append_trace(target_go, row=2, col=1)
fig.add_trace(
go.Scatter(x=tmp.index, y=tmp["confirmed shifted on 5"], name="shift=5"),
row=2, col=1
)
fig.add_trace(
go.Scatter(x=tmp.index, y=tmp["confirmed shifted on 12"], name="shift=12"),
row=2, col=1
)
fig.update_layout(
autosize=True,
# width=1000,
height=700,
margin=dict(
l=0,
r=0,
b=0,
t=30,
pad=4
)
)
path = os.path.join(plotly_save_to, "covid19.html")
fig.write_html(path) # fig.show()
display(HTML(path))
Найденный сдвиг: 5
Оба графика довольно сильно осциллируют. Применим метод скользящего среднего по неделе, чтобы избавиться от наблюдаемого “шума”. Для этого воспользуемся методом pd.DataFrame.rolling, который предназначен для вычислений с скользящим окном.
smoothed_daily_df = daily_df.rolling(7).mean()
fig = px.line(smoothed_daily_df)
fig.write_html(plotly_tmp) # fig.show()
display(HTML(plotly_tmp))
Данные о коронавирусе в России от яндекса¶
Первый 5 строк файла выглядят так.
"Дата","Регион","Заражений","Выздоровлений","Смертей","Смертей за день","Заражений за день","Выздоровлений за день"
20.09.2020,Томская обл.,6775,5554,79,1,62,92
26.09.2020,Костромская обл.,4553,3329,67,5,54,44
02.09.2021,Ямало-Ненецкий АО,47772,45810,625,4,102,121
15.12.2020,Сахалинская обл.,13406,10292,11,0,142,366
Видно, что в столбце “Дата” хранятся даты в формате DD.MM.YYYY, который не является стандартным для pandas. Чтобы корректно прочитать эти данные, определим функцию, которая преобразует строку с датой в формате DD.MM.YYYY в datetime объект и при чтении файла передадим эту функцию в качестве парсера дат.
import os
import pandas as pd
from datetime import datetime
def date_parser(x):
return datetime.strptime(x, r"%d.%m.%Y")
folder = "covid19"
filename = "yandex_data.csv"
path = os.path.join("covid19", "yandex_data.csv")
df = pd.read_csv(path, parse_dates=["Дата"], index_col="Дата", date_parser=date_parser)
df.info()
<class 'pandas.core.frame.DataFrame'>
DatetimeIndex: 52938 entries, 2020-09-20 to 2021-04-20
Data columns (total 7 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Регион 52938 non-null object
1 Заражений 52938 non-null int64
2 Выздоровлений 52938 non-null int64
3 Смертей 52938 non-null int64
4 Смертей за день 52938 non-null int64
5 Заражений за день 52938 non-null int64
6 Выздоровлений за день 52938 non-null int64
dtypes: int64(6), object(1)
memory usage: 3.2+ MB
Что мы видим:
53938 строк;
7 полностью заполненных столбцов;
даты от 20 сентября 2021 года до 20 апреля 2021 года в качестве индекса;
Посмотрим первые 5 строк таблицы.
df.head()
| Регион | Заражений | Выздоровлений | Смертей | Смертей за день | Заражений за день | Выздоровлений за день | |
|---|---|---|---|---|---|---|---|
| Дата | |||||||
| 2020-09-20 | Томская обл. | 6775 | 5554 | 79 | 1 | 62 | 92 |
| 2020-09-26 | Костромская обл. | 4553 | 3329 | 67 | 5 | 54 | 44 |
| 2021-09-02 | Ямало-Ненецкий АО | 47772 | 45810 | 625 | 4 | 102 | 121 |
| 2020-12-15 | Сахалинская обл. | 13406 | 10292 | 11 | 0 | 142 | 366 |
| 2020-09-21 | Волгоградская обл. | 14158 | 12376 | 131 | 0 | 95 | 13 |
Можно заметить, что таблица не упорядоченна по индексу. Отсортируем её, для нашего удобства.
df.sort_index(inplace=True)
df.head()
| Регион | Заражений | Выздоровлений | Смертей | Смертей за день | Заражений за день | Выздоровлений за день | |
|---|---|---|---|---|---|---|---|
| Дата | |||||||
| 2020-03-12 | Сахалинская обл. | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-03-12 | Северная Осетия | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-03-12 | Камчатский край | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-03-12 | Липецкая обл. | 3 | 0 | 0 | 0 | 3 | 0 |
| 2020-03-12 | Крым | 0 | 0 | 0 | 0 | 0 | 0 |
Кстати, можно заметить, что в начало попали строки от 4 января 2020 года, что гораздо раньше диапазона дат, полученного в команде df.info.
df.info()
fig = px.line(df, y=["Заражений за день"], color="Регион", title="Заражений за день")
fig.write_html(plotly_tmp) # fig.show()
display(HTML(plotly_tmp))
<class 'pandas.core.frame.DataFrame'>
DatetimeIndex: 52938 entries, 2020-03-12 to 2021-11-24
Data columns (total 7 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Регион 52938 non-null object
1 Заражений 52938 non-null int64
2 Выздоровлений 52938 non-null int64
3 Смертей 52938 non-null int64
4 Смертей за день 52938 non-null int64
5 Заражений за день 52938 non-null int64
6 Выздоровлений за день 52938 non-null int64
dtypes: int64(6), object(1)
memory usage: 3.2+ MB
Т.е. в таблице данные представлены от 4 января 2020 года до 24 ноября 2021 года.
Т.к. python работает с unicode, то можно оставить имена столбцов и на русском языке, но в целях демонстрации переименуем столбцы на английский язык методом DataFrame.rename. На вход он принимает словарь, в котором ключи — старые названия столбцов, значения — новые. По умолчанию этот метод возвращает копию таблицы с новыми столбцами имен. Изменить это можно параметром inplace=True.
Индекс переименовывается отдельно.
df.rename({
"Регион": "region",
"Заражений": "cases",
"Выздоровлений": "recoveries",
"Смертей": "deaths",
"Смертей за день": "deaths per day",
"Заражений за день": "cases per day",
"Выздоровлений за день": "recoveries per day"
}, inplace=True, axis="columns")
df.index.rename("date", inplace=True)
df
| region | cases | recoveries | deaths | deaths per day | cases per day | recoveries per day | |
|---|---|---|---|---|---|---|---|
| date | |||||||
| 2020-03-12 | Сахалинская обл. | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-03-12 | Северная Осетия | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-03-12 | Камчатский край | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-03-12 | Липецкая обл. | 3 | 0 | 0 | 0 | 3 | 0 |
| 2020-03-12 | Крым | 0 | 0 | 0 | 0 | 0 | 0 |
| ... | ... | ... | ... | ... | ... | ... | ... |
| 2021-11-24 | Владимирская обл. | 70566 | 54233 | 2432 | 9 | 318 | 131 |
| 2021-11-24 | Курганская обл. | 43855 | 34847 | 854 | 6 | 207 | 122 |
| 2021-11-24 | Волгоградская обл. | 115111 | 98848 | 4360 | 37 | 458 | 381 |
| 2021-11-24 | Красноярский край | 148381 | 132019 | 7286 | 33 | 710 | 695 |
| 2021-11-24 | Еврейская АО | 10330 | 8862 | 327 | 0 | 42 | 51 |
52938 rows × 7 columns
Сравним статистику по регионам. Для этого выберем последней день данных и построим круговую диаграмму заболеваемости по регионам.
last_day = df.index.max()
last_day_df = df.loc[last_day]
fig = px.pie(last_day_df, values="cases", names='region')
fig.update_traces(textposition="inside")
fig.write_html(plotly_tmp) # fig.show()
display(HTML(plotly_tmp))
last_day_df
| region | cases | recoveries | deaths | deaths per day | cases per day | recoveries per day | |
|---|---|---|---|---|---|---|---|
| date | |||||||
| 2021-11-24 | Ингушетия | 25647 | 23801 | 414 | 3 | 83 | 90 |
| 2021-11-24 | Пензенская обл. | 89547 | 61771 | 4097 | 19 | 391 | 291 |
| 2021-11-24 | Вологодская обл. | 87778 | 77468 | 2499 | 16 | 397 | 417 |
| 2021-11-24 | Московская обл. | 577907 | 509872 | 10116 | 56 | 1842 | 2180 |
| 2021-11-24 | Ленинградская обл. | 88124 | 68927 | 2747 | 10 | 425 | 435 |
| ... | ... | ... | ... | ... | ... | ... | ... |
| 2021-11-24 | Владимирская обл. | 70566 | 54233 | 2432 | 9 | 318 | 131 |
| 2021-11-24 | Курганская обл. | 43855 | 34847 | 854 | 6 | 207 | 122 |
| 2021-11-24 | Волгоградская обл. | 115111 | 98848 | 4360 | 37 | 458 | 381 |
| 2021-11-24 | Красноярский край | 148381 | 132019 | 7286 | 33 | 710 | 695 |
| 2021-11-24 | Еврейская АО | 10330 | 8862 | 327 | 0 | 42 | 51 |
85 rows × 7 columns
Диаграмма получилась очень загроможденной. Продемонстрируем работу метода pd.unique и посчитаем количество регионов, которые встречаются в таблице.
unique_regions = df["region"].unique()
print(f"{len(unique_regions)} регионов, среди которых")
print(np.sort(unique_regions))
85 регионов, среди которых
['Адыгея' 'Алтай' 'Алтайский край' 'Амурская обл.' 'Архангельская обл.'
'Астраханская обл.' 'Башкортостан' 'Белгородская обл.' 'Брянская обл.'
'Бурятия' 'Владимирская обл.' 'Волгоградская обл.' 'Вологодская обл.'
'Воронежская обл.' 'Дагестан' 'Еврейская АО' 'Забайкальский край'
'Ивановская обл.' 'Ингушетия' 'Иркутская обл.' 'Кабардино-Балкария'
'Калининградская обл.' 'Калмыкия' 'Калужская обл.' 'Камчатский край'
'Карачаево-Черкессия' 'Карелия' 'Кемеровская обл.' 'Кировская обл.'
'Коми' 'Костромская обл.' 'Краснодарский край' 'Красноярский край' 'Крым'
'Курганская обл.' 'Курская обл.' 'Ленинградская обл.' 'Липецкая обл.'
'Магаданская обл.' 'Марий Эл' 'Мордовия' 'Москва' 'Московская обл.'
'Мурманская обл.' 'Ненецкий АО' 'Нижегородская обл.' 'Новгородская обл.'
'Новосибирская обл.' 'Омская обл.' 'Оренбургская обл.' 'Орловская обл.'
'Пензенская обл.' 'Пермский край' 'Приморский край' 'Псковская обл.'
'Ростовская обл.' 'Рязанская обл.' 'Самарская обл.' 'Санкт-Петербург'
'Саратовская обл.' 'Саха (Якутия)' 'Сахалинская обл.' 'Свердловская обл.'
'Севастополь' 'Северная Осетия' 'Смоленская обл.' 'Ставропольский край'
'Тамбовская обл.' 'Татарстан' 'Тверская обл.' 'Томская обл.'
'Тульская обл.' 'Тыва' 'Тюменская обл.' 'Удмуртия' 'Ульяновская обл.'
'ХМАО – Югра' 'Хабаровский край' 'Хакасия' 'Челябинская обл.' 'Чечня'
'Чувашия' 'Чукотский АО' 'Ямало-Ненецкий АО' 'Ярославская обл.']
В таблице присутствуют данные по всем 85 регионам РФ.
85 субъектов Федерации: 22 республики, 9 краев, 46 областей, 3 города федерального значения, 1 автономная область, 4 автономных округа.
Для простоты изложения выберем 3 региона, с наибольшим количеством заболевших за весь период времени, и 2 региона с наименьшим количеством заболевших.
Сначала найдем такие регионы. Для этого воспользуемся методами DataFrame.nlargest и DataFrame.nsmallest, которые позволяют отобрать n строк таблицы с наибольшими значениями в нужном столбце (или в столбцах). В таблице last_day_df в столбце cases как раз хранятся нужные нам значения по регионам. Применив эти два метода к этой таблице, получаем таблицу из нужных нам строк. Нам нужны только регионы, то извлекаем из этой таблицы столбец region.
Объединить два полученных столбца в один можно методом pd.concat. Т.к. столбцы необходимо совместить “друг под другом”, то в axis указываем index. Индекс этих столбцов берется из таблицы исходной таблицы last_day_df, т.е. это индекс с одной и той же датой. Не учитывать такой индекс при объедении можно указав параметр ignore_index=True.
largest_regions = last_day_df.nlargest(3, columns=["cases"])["region"]
smallest_regions = last_day_df.nsmallest(2, columns=["cases"])["region"]
regions = pd.concat([largest_regions, smallest_regions], axis="index", ignore_index=True)
print(regions)
0 Москва
1 Санкт-Петербург
2 Московская обл.
3 Чукотский АО
4 Ненецкий АО
Name: region, dtype: object
Теперь из исходной таблицы можно отобрать строки с нужными регионами, проверяя значение в столбце region, т.е. методом логического отбора. Удобно использовать здесь метод isin.
small_df = df[
df["region"].isin(regions)
].copy()
small_df
| region | cases | recoveries | deaths | deaths per day | cases per day | recoveries per day | |
|---|---|---|---|---|---|---|---|
| date | |||||||
| 2020-03-12 | Москва | 21 | 0 | 0 | 0 | 21 | 0 |
| 2020-03-12 | Московская обл. | 4 | 0 | 0 | 0 | 4 | 0 |
| 2020-03-12 | Санкт-Петербург | 1 | 0 | 0 | 0 | 1 | 0 |
| 2020-03-13 | Московская обл. | 5 | 0 | 0 | 0 | 1 | 0 |
| 2020-03-13 | Санкт-Петербург | 4 | 0 | 0 | 0 | 3 | 0 |
| ... | ... | ... | ... | ... | ... | ... | ... |
| 2021-11-24 | Московская обл. | 577907 | 509872 | 10116 | 56 | 1842 | 2180 |
| 2021-11-24 | Чукотский АО | 2793 | 2406 | 24 | 0 | 10 | 15 |
| 2021-11-24 | Москва | 1926701 | 1745284 | 33635 | 93 | 2533 | 5393 |
| 2021-11-24 | Санкт-Петербург | 775795 | 709724 | 25802 | 73 | 1387 | 3147 |
| 2021-11-24 | Ненецкий АО | 3145 | 2865 | 72 | 0 | 8 | 39 |
3105 rows × 7 columns
Построим график заражений только для отобранных регионов.
fig = px.line(small_df, y="cases per day", color="region")
fig.write_html(plotly_tmp) # fig.show()
display(HTML(plotly_tmp))
На глаз заметны волны. Кроме того, кажется, что эти волны происходят почти синхронно. Вычислим взаимные корреляции этих графиков. Для этого удобно использовать метод DataFrame.correlate, который вычисляет корреляцию между всеми столбцами таблицы. Создадим таблицу с нужными нам столбцами.
data = {}
for region in regions:
col = small_df[small_df["region"] == region]["cases per day"]
data[region] = col.copy()
to_correlate = pd.DataFrame(data)
to_correlate
| Москва | Санкт-Петербург | Московская обл. | Чукотский АО | Ненецкий АО | |
|---|---|---|---|---|---|
| date | |||||
| 2020-03-12 | 21 | 1 | 4 | NaN | NaN |
| 2020-03-13 | 5 | 3 | 1 | NaN | NaN |
| 2020-03-14 | 9 | 1 | 1 | NaN | NaN |
| 2020-03-15 | 0 | 0 | 3 | NaN | NaN |
| 2020-03-16 | 18 | 3 | -1 | NaN | NaN |
| ... | ... | ... | ... | ... | ... |
| 2021-11-20 | 3239 | 2637 | 1885 | 25.0 | 16.0 |
| 2021-11-21 | 3438 | 2496 | 1909 | 10.0 | 16.0 |
| 2021-11-22 | 2786 | 2215 | 1784 | 11.0 | 16.0 |
| 2021-11-23 | 2749 | 1060 | 1871 | 10.0 | 14.0 |
| 2021-11-24 | 2533 | 1387 | 1842 | 10.0 | 8.0 |
623 rows × 5 columns
corr = to_correlate.corr()
fig = px.imshow(corr, zmin=-1, zmax=1)
fig.write_html(plotly_tmp) # fig.show()
display(HTML(plotly_tmp))
corr
| Москва | Санкт-Петербург | Московская обл. | Чукотский АО | Ненецкий АО | |
|---|---|---|---|---|---|
| Москва | 1.000000 | 0.678963 | 0.691879 | 0.357444 | 0.349546 |
| Санкт-Петербург | 0.678963 | 1.000000 | 0.724287 | 0.576447 | 0.511084 |
| Московская обл. | 0.691879 | 0.724287 | 1.000000 | 0.680546 | 0.639753 |
| Чукотский АО | 0.357444 | 0.576447 | 0.680546 | 1.000000 | 0.757208 |
| Ненецкий АО | 0.349546 | 0.511084 | 0.639753 | 0.757208 | 1.000000 |
